Platform Technology
Platform Technologies
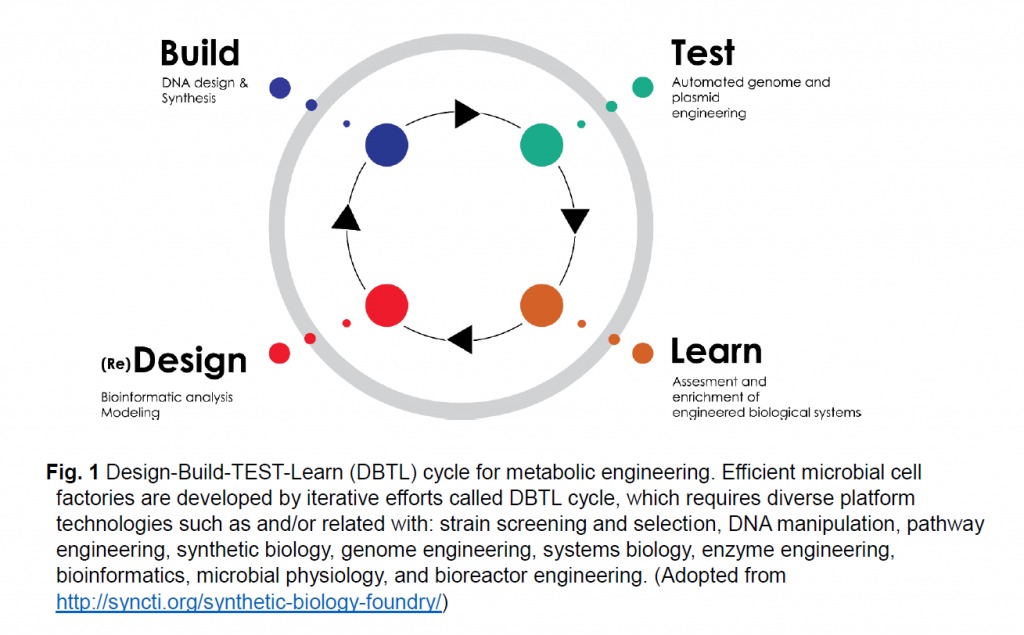
Development of industrial microorganisms for biorefinery is challenging and time-consuming. It requires iterative efforts called ‘Design-Build-TEST-Learn (DBTL)’ cycle. First, in the ‘Design’ step, a biological system or metabolic pathways should be designed to synthesize a desired molecules via coding of these pathways into DNA parts and assembly instructions. Second, in the ‘Build’ step, the biological system should be built from DNA parts and production-relevant microbial chassis, using inputs from ‘Design’ and tools developed through synthetic biology. Third, in the Test step, the engineered biological system is tested to determine if and how the system carries out the desired function, with the help of cell physiology, omics and systems biology. Finally, in the ‘Learn’ step, information on the performance of the engineered biological systems are collected, examined and analyzed. To improve the performance of biological systems, further D-B-T-L cycles are followed. In D-B-T-L cycles, many platform technologies, specific for each microbial cell factory should be developed and used.
Screening and selection of proper host strains:
Before building or engineering metabolic pathway to produce a desired molecule, appropriate strain should be carefully selected. Not only the use of known microorganisms but also the screening of new isolates are considered. Many technological criteria are examined as follows: growth potential or growth requirement for nutrients and environmental conditions, inherent metabolic pathways related with cellular growth and metabolites production, presence and/or regulation of expression of important enzymes, regeneration of cofactors, tolerance to toxic intermediates and target products, bioreactor operational conditions, etc. Currently, we are studying many bacteria including Escherichia coli, Clostridium, Pseudomonas, Klebsiella, Corynebacterium, Citrobacter, Bacillus, Lactobacillus, and Brevibacterium.
Gene manipulation technology:
For selected microorganisms, we develop and use diverse gene manipulation technologies for knock-out or knock-in of genes, and alteration of gene expression. Gene manipulations are performed at both episomal and genomic levels. At the genome level, in addition to the conventional Lamda-Red method and pop-in/pop-out method, the newly developed CRISPR-Cas9 method, MAGE (Multiplex Automated Genome Engineering) method targeting many genes simultaneously, and/or CRIMAGE (Combination of CRISPR-Cas9 and MAGE) are being developed and employed for efficient genome engineering.
Metabolic engineering:
To optimize the production of a target molecule in the selected microorganisms, it is necessary to modify metabolic pathways by deleting the existing enzymes/pathways and introducing new enzymes/pathways. Further, we can re-design the biological system by using novel pathways or mutating global regulatory proteins and/or their expression. For quantitative analysis of the biological system, we adopt mathematical cell growth model based on flux balance analysis (FBA). For systems approaches, multi-omics analyses are also integrated. For screening of the optimal strains showing the desired traits, it is necessary to develop and use efficient screening systems based on biosensors.
Synthetic biology:
It is a technology to synthesize an expression system that can easily control gene expression or gene expression level in various combinations for introduction of new pathway or optimal expression of pathway. We have developed and used individual or complex gene cassettes required for pathway synthesis for important pathways involved in the biosynthesis of 3-HP, 1,3-PDO, and lactone compounds, which our laboratory is focusing on.
Enzyme engineering:
In the development of microbial catalysts that convert sugar or glycerol feedstocks to the desired end products, the characterization and improvement of relevant enzymes are critically important. In many cases, it is necessary to modify the kinetic characteristics of enzymes, such as maximum reaction rate, stability to the substrate and other intermediates, feedback inhibition by product, temperature or pH dependency, and selectivity to substrate and coenzyme. Determination of crystal structure, identification of active sites and docking between enzymes and substrates or cofactors are conducted to reveal biochemical characteristics and mutation targets of enzymes. Site-directed mutagenesis and directed evolution are followed to develop enzyme mutant libraries which are applied to high-throughput screening (HTS) systems.
Bioinformatics:
In the search for proper enzymes, genes, metabolic pathways and even microorganisms, using known and readily-available information as much as possible is essential to improve the efficiency and speed of development of microbial and enzyme catalyst. In order to select optimal microorganisms, bioinformatics is actively used to compare microbial genomic information. Further, bioinformatics is an indispensable tool to investigate the sequence of genes or proteins, to interpret the structures of enzymes, to synthesize metabolic pathways, and to determine the pathway of conversion.
Bioreactor engineering:
Microbial catalysts developed at the flask level should be verified at the bench-scale bioreactor and, furthermore, performance should be assured in the pilot bioreactor level. During culture experiments and scale-up process, low-cost media and operating conditions must be established. In addition, bioreactor operation should be combined with the separation and purification process. Currently, bioreactor operation and scale-up are jointly-studied with collaborating companies.
기반기술 (Platform Technology)
바이오리파이너리를 위한 상업용 미생물의 개발은 까다롭고 시간이 많이 소요됩니다. 'Design-Build-TEST-Learn (DBTL)' 사이클을 통한 반복적인 노력이 필요합니다.
첫째, 'Design (디자인)' 단계에서는 생물학적 시스템이나 대사 경로를 설계하게 되는데 이러한 경로를 DNA 부품과 어셈블리를 가능하게 하는 로직으로 코딩합니다. 둘째, 'Build' 단계에서는 합성 생물학을 통해 개발된 '디자인' 및 도구의 입력을 사용하여 DNA 부품 및 생산 관련 미생물 섀시를 만들고 이를 미생물 내로 도입하게 됩니다. 셋째, ‘Test (시험)’ 단계에서는, 설계된 생물학적 시스템을 시험하여 세포 생리학, 오믹스 및 시스템 생물학의 도움을 받아 시스템이 원하는 기능을 수행하는지 여부를 결정하고 어떻게 기능하는지 조사합니다. 마지막으로 ‘Learn (학습)’ 단계에서는 개발된 생물 시스템의 성능에 대한 정보를 수집, 검사 및 분석합니다.
생물학적 시스템의 성능을 향상시키기 위해, 그리고 만족할 만한 결과를 얻을 때까지 D-B-T-L 사이클을 반복적으로 진행합니다. D-B-T-L 사이클에서는 각 미생물 세포 공장에 특화된 많은 플랫폼 기술을 개발하여 사용해야 하며, 다음과 같은 기술이 연구/사용되고 있습니다.
- 균주 선별 기술: 기존 알려진 균주는 물론 자연으로부터 새로운 균주를 찾아내고 선별하며 적절한 균주를 목적에 맞도록 선택함. 균주는 사용하려는 원료물질은 물론, 균주의 성장 특성, 원료물질 전환 대사경로, 효소의 발현 기작, 효소의 활성, 조효소 재생, 생산물의 독성, 생물반응기 운전조건 등을 고려하여 선발하게 됨. 현재 대장균, Clostridium, Pseudomonas, Klebsiella, Corynebacterium, Citrobacter, Bacillus, Lactobacillus, Brevibacterium 등이 연구되고 있음.
- 유전자 조작기술 (UNIST 이성국 교수님과 협업): 선택된 균주 내 특정 유전자를 제거하거나 새로운 유전자를 발현, 혹은 유전자의 발현 정도를 조절하기 위하여 사용하는 기술임. 전통적인 lamda-red 방법은 물론 흔적을 남기지 않는 pop-in pop-out 방법, genome 상의 유전자 변형을 위한 유전자 가위(CRISPR-Cas9), 여러 유전자를 동시에 변형시킬 수 있는 MAGE (Multiplex Automated Genome Engineering) 등을 사용하고 있음. (UNIST 이성국 교수님 연구실과 협업)
- 대사공학 기술 (UNIST 이성국 교수님, POSTECH 정규열 교수님과 협업): 원하는 최종산물의 최적 생산을 위하여 미생물의 대사 경로를 변경하는 것으로 대상이 되는 균주 내 기존의 대사 경로를 제거하거나 변형하고 새로운 대사경로를 도입함. 더 나아가 새로운 반응 특성을 갖는 효소를 이용하거나 세포 전체 유전자 발현을 제어하는 제어 인자의 변형을 통해 시스템 수준에서 중요한 대사 경로를 재설계하게 됨. 탄소원이 이용되는 경로를 조사하기 위하여 대사흐름을 수학적 혹은 실험적으로 분석하고 이를 대사산물의 최적생산에 이용함.
- 합성생물학 기술 (UNIST 이성국 교수님 연구실, POSTECH 정규열 교수님 연구실과 협업): 새로운 경로를 도입하거나 경로의 최적 발현을 위하여 중요한 유전자의 발현이나 유전자 발현 수준 등을 여러 조합으로 쉽게 조절할 수 있도록 발현 시스템을 합성하는 기술임. 본 연구실이 중점적으로 연구하는 3-HP, 1,3-PDO, 락톤화합물 등의 생합성에 관여하는 중요한 경로에 대하여 경로합성에 필요한 개별 혹은 복합유전자 카세트를 개발하고 이용하고 있음.
- 효소공학기술 (UNIST 김용환 교수님, 아주대 유태현 교수님, 경북대 김경진 교수님, 부산대 이선구 교수님과 협업): 당이나 글리세롤 원료를 원하는 최종 산물로 전환하는 미생물 촉매 개발에 있어 효소의 특성, 즉 활성이나 안정성, 발현 용이성 등은 가장 중요한 연구 대상임. 특히 효소의 동역학적 특성인 최대 반응속도, 기질에 대한 안정성, 생성물 되먹임 방해, 온도나 pH 의존성, 기질과 조효소에 대한 선택성 등을 원하는 수준으로 변화시키는 것이 필요함. 효소의 구조를 밝히고 이에 기반한 site-directed mutagenesis, directed evolution, 그리고 이를 통해 제작된 큰 규모의 변이 효소 library에서 원하는 효소를 선택적으로 골라내는 high throughput screening (HTS) 기술 등이 개발되었고 이용되고 있음.
- 바이오 인포메틱스 (생물 정보학): 효소나 유전자, 그리고 대사경로를 발굴하는데 있어 이미 알려진 여러 정보를 이용하는 것이 미생물이나 효소 촉매개발의 효율성과 속도를 높이는데 필수적임. 최적 미생물을 선정하기 위하여 미생물 유전체 정보를 비교하거나 유전자나 단백질의 서열을 조사하는 것은 물론 효소의 구조에 대한 해석, 대사 경로의 합성이나 전환 경로의 결정하기 위하여 생물 정보학을 적극 활용하고 있음.
- 생물반응기 기술 (노루홀딩스 및 GS Caltex와 협업): 플라스크 수준에서 개발된 미생물 촉매는 벤치 규모 생물반응기 수준에서 성능이 확인되고 더 나아가 pilot, 공장 규모 생물반응기에서 성능이 보장되어야 함. 더 나아가 저렴한 배지 조성과 운전조건이 확립되어야 하고 분리정제 공정과 결합될 수 있어야 함. 이를 위해 생물 반응기 운전, 설계 기술이 필요하며 이를 위하여 기업체와 공동연구를 수행하고 있음.
